Seurat单细胞处理流程之八:pySCENIC转录因子分析
简介
这一步要转为python进行分析,因为R版本的scenic只能调用大核心,导致速度非常慢,经常是三四天的运行时间,为了效率,这里使用python版本的scenic进行转录因子预测。
1.运行前准备
运行pyscenic前需要准备以下几个文件: 1:下载motif排序文件,motif 注释文件和转录因子文件 以人类基因组hg19版本为参考,转录起始位点(TSS)上下游5kb区域的基因与基序(motifs)的排名数据,数据整合7个物种信息用于评估基因与基序结合可能性,确定转录因子和靶基因间调控关系 wget https://resources.aertslab.org/cistarget/databases/homo_sapiens/hg19/refseq_r45/mc9nr/gene_based/hg19-tss-centered-5kb-7species.mc9nr.genes_vs_motifs.rankings.feather 2:基序到转录因子的映射关系,通过分析转录因子结合位点基序,识别可能结合特定基序的转录因子。 wget https://resources.aertslab.org/cistarget/motif2tf/motifs-v10nr_clust-nr.hgnc-m0.001-o0.0.tbl 3:人类基因组中所有转录因子列表 wget https://resources.aertslab.org/cistarget/tf_lists/allTFs_hg38.txt
这几个文件有几个坑需要注意一下,否则对应上的转录因子数量会特别少。 坑1:motif排序文件与人类基因组中所有转录因子列表分为hg19和hg38版本,一定要用相同的版本。hg19版本的转录因子列表在:https://github.com/aertslab/pySCENIC/blob/master/resources/hs_hgnc_tfs.txt 坑2:基序到转录因子的映射关系里v8, v9, v10 是 motif 集合的版本号,每个版本基于不同的研究和数据库更新。最新的 v10 版本基于 2022 年的 SCENIC+ motif 集合。
2.环境配置
# pyscenic环境创建流程
# conda env remove --name pyscenic
# conda create -n pyscenic python=3.8.1
# conda activate pyscenic
# !!!!!按照下面的顺序安装,不然可能出现彼此不兼容的报错
# 参考:https://www.jianshu.com/p/dc7397fda327
# pip install numpy==1.19.5
# pip install pandas==1.3.5
# pip install numba==0.56.4
# pip install pyscenic==0.12.1
# 检查安装软件版本
# python -c "import numpy; print(numpy.__version__)"
# python -c "import pandas; print(pandas.__version__)"
# python -c "import pyscenic; print(pyscenic.__version__)"
# pyscenic -h
3. pbmc的转录因子分析
## 加载模块
from pathlib import Path
import numpy as np
import pandas as pd
import scanpy as sc
import matplotlib.pyplot as plt
import loompy
import os
OMP: Info #276: omp_set_nested routine deprecated, please use omp_set_max_active_levels instead.
# 获取当前目录
os.getcwd()
os.listdir(os.getcwd())
# 1. 设置工作目录
os.chdir("/mnt/DEV_8T/zhaozm/seurat全流程/pyscenic/")
OUTPUT_DIR = "/mnt/DEV_8T/zhaozm/seurat全流程/pyscenic/data"
Path(OUTPUT_DIR).mkdir(parents=True, exist_ok=True)
## 读取示例文件
adata = sc.read_h5ad("../../scanpy_单细胞流程/pbmc_test/adata_注释后.h5ad")
## 检查示例文件
adata
AnnData object with n_obs × n_vars = 2638 × 1838
obs: 'n_genes', 'n_genes_by_counts', 'total_counts', 'total_counts_mt', 'pct_counts_mt', 'leiden', 'celltype'
var: 'gene_ids', 'n_cells', 'mt', 'n_cells_by_counts', 'mean_counts', 'pct_dropout_by_counts', 'total_counts', 'ribo', 'hb', 'highly_variable', 'means', 'dispersions', 'dispersions_norm', 'mean', 'std'
uns: 'celltype_colors', 'hvg', 'leiden', 'leiden_colors', 'leiden_sizes', 'log1p', 'neighbors', 'paga', 'pca', 'rank_genes_groups', 'umap'
obsm: 'X_pca', 'X_umap'
varm: 'PCs'
obsp: 'connectivities', 'distances'
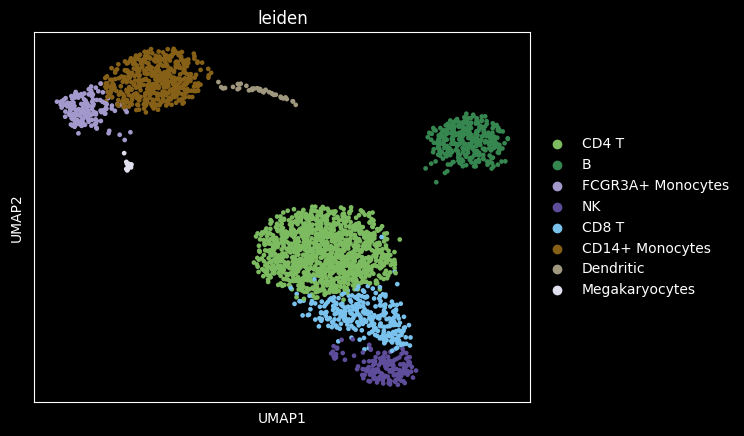
## 绘制umap图
sc.pl.umap(adata,color=["leiden"])
print(adata.raw) # 如果是 None,说明没有保存原始数据
Raw AnnData with n_obs × n_vars = 2638 × 13714
var: 'gene_ids', 'n_cells', 'mt', 'n_cells_by_counts', 'mean_counts', 'pct_dropout_by_counts', 'total_counts', 'ribo', 'hb', 'highly_variable', 'means', 'dispersions', 'dispersions_norm'
## 转换为loom文件
adata_raw = adata.raw.to_adata()
rownames = dict(Gene=adata_raw.var_names.tolist())
colnames = dict(CellID=adata_raw.obs_names.tolist())
loompy.create(filename=os.path.join(OUTPUT_DIR, "pbmc.loom"), layers=adata_raw.X.transpose(), row_attrs=rownames, col_attrs=colnames)
# ## 接下来的运行转到bash脚本里运行
# #!/bin/bash
# # 设置数据库目录路径
# dir=/mnt/DEV_8T/zhaozm/seurat全流程/pyscenic/data/
# tfs=$dir/hs_hgnc_tfs.txt
# feather=$dir/hg19-tss-centered-10kb-10species.mc9nr.genes_vs_motifs.rankings.feather
# tbl=$dir/motifs-v9-nr.hgnc-m0.001-o0.0.tbl
# # 确保数据库文件完整无误
# ls $tfs $feather $tbl
# ######################
# # 运行pySCENIC for CD4
# ######################
# input_loom=$dir/pbmc.loom
# # 2.1 GRN推断(推测基因调控网络)
# pyscenic grn \
# --num_workers 12 \
# --output adj.pbmc.tsv \
# --method grnboost2 \
# $input_loom \
# $tfs
# # 2.2 cisTarget(使用Motif和Rankings进行基因调控网络修饰)
# pyscenic ctx \
# adj.pbmc.tsv $feather \
# --annotations_fname $tbl \
# --expression_mtx_fname $input_loom \
# --mode "dask_multiprocessing" \
# --output reg.pbmc.csv \
# --num_workers 12
# pyscenic ctx \
# # 2.3 AUCell(计算每个细胞中的转录因子活性)
# pyscenic aucell \
# $input_loom \
# reg.pbmc.csv \
# --output out_pbmc_SCENIC.loom
最后,脚本挂到后台运行即可 nohup bash scenic.bash &